食管癌是全球高发的恶性肿瘤,我国食管癌发病率和死亡率却均居世界首位,最新统计数据显示,在中国每年约有48万新增食管癌病例,37万例食管癌患者死亡【1】。在中国食管鳞癌(ESCC)是主要的病理类型,其预后差、恶性程度高,是一类治愈率极低的肿瘤。近年来随着技术的发展,对食管鳞癌组织的基因检测已有近2000多例【2-4】,基因变异的致病机理及调控网络已呈现在人们的视野,但其观察视角单一,且治疗方案并未因此得到改善,目前除了传统治疗方式外,缺乏精准的治疗手段。
为阐明ESCC的致病机制及分子分型,寻找诊断标志物、药物靶点和制定临床治疗方案,刘芝华/詹启敏/崔永萍团队前期对我国食管鳞癌患者样本进行了508对全基因组测序和94例蛋白质组及磷酸化组测序【2,5】,结果表明, ESCC是一种由于基因组大规模重组和拷贝数扩增缺失造成的癌症,基因组的不稳定性和重组导致患者之间存在显著的异质性;TCGA团队研究的数据已发表食管腺癌(欧美国家高发的食管癌亚型)与食管鳞癌测序结果比对【6】,结果发现食管鳞癌与食管腺癌是完全不同的肿瘤,须采用不同的治疗方式。而从多组学多层面多视角挖掘ESCC的致病机制,明确其分子分型,提其高诊治水平,已成为食管鳞癌研究领域的重大研究课题。
2022年12月29日,中国医学科学院肿瘤医院刘芝华/焦宇辰团队、深圳湾实验室詹启敏/崔永萍团队合作(中国医学科学院肿瘤医院刘芝华教授、助理研究员赵丫卉、博士后刘雨豪、主任医师黄镜、博士生韦文青和山西医科大学副教授孔鹏洲、主任医师徐恩伟为本文共同第一作者)在Cancer Cell上发表了文章Integrated Multi-omics Profiling Yields a Clinically-Relevant Molecular Classification for Esophageal Squamous Cell Carcinoma【7】。研究者基于多组学数据分析,将食管鳞癌分为四个分子亚型:细胞周期通路激活型(CCA)、NRF2通路激活型(NRFA)、免疫抑制型(IS)和免疫调节型(IM)。这是国际上首次基于多组学数据的食管鳞癌分类标准,并为每个亚型寻找了潜在的治疗靶标和/或诊断标志物。

研究者纳入155例食管鳞癌患者配对样本进行了全基因组、表观基因组、转录组和蛋白质组数据层面的全面整合分析;利用多组学分析首次将食管鳞癌分为CCA、NRFA、IS和IM四个亚型(图1)。CCA亚型体现在细胞周期检查点基因的变异,而NRFA亚型则有NRF2通路基因的激活。CCA和NRFA亚型患者均具有CpG岛甲基化表型(E-CIMP+),并与淋巴节转移和临床分期相关。IS和IM亚型是免疫细胞高浸润型,但肿瘤组织浸润的免疫细胞类型不同。研究者进一步利用43例接受PD-1抗体单药治疗队列研究,发现IM亚型对免疫检查点阻断疗法(ICB)有更好的治疗响应,并基于该队列ESCC组织多组学数据开发了识别IM亚型患者的分子特征分类器,体现了基于多组学数据分子分型的临床价值。
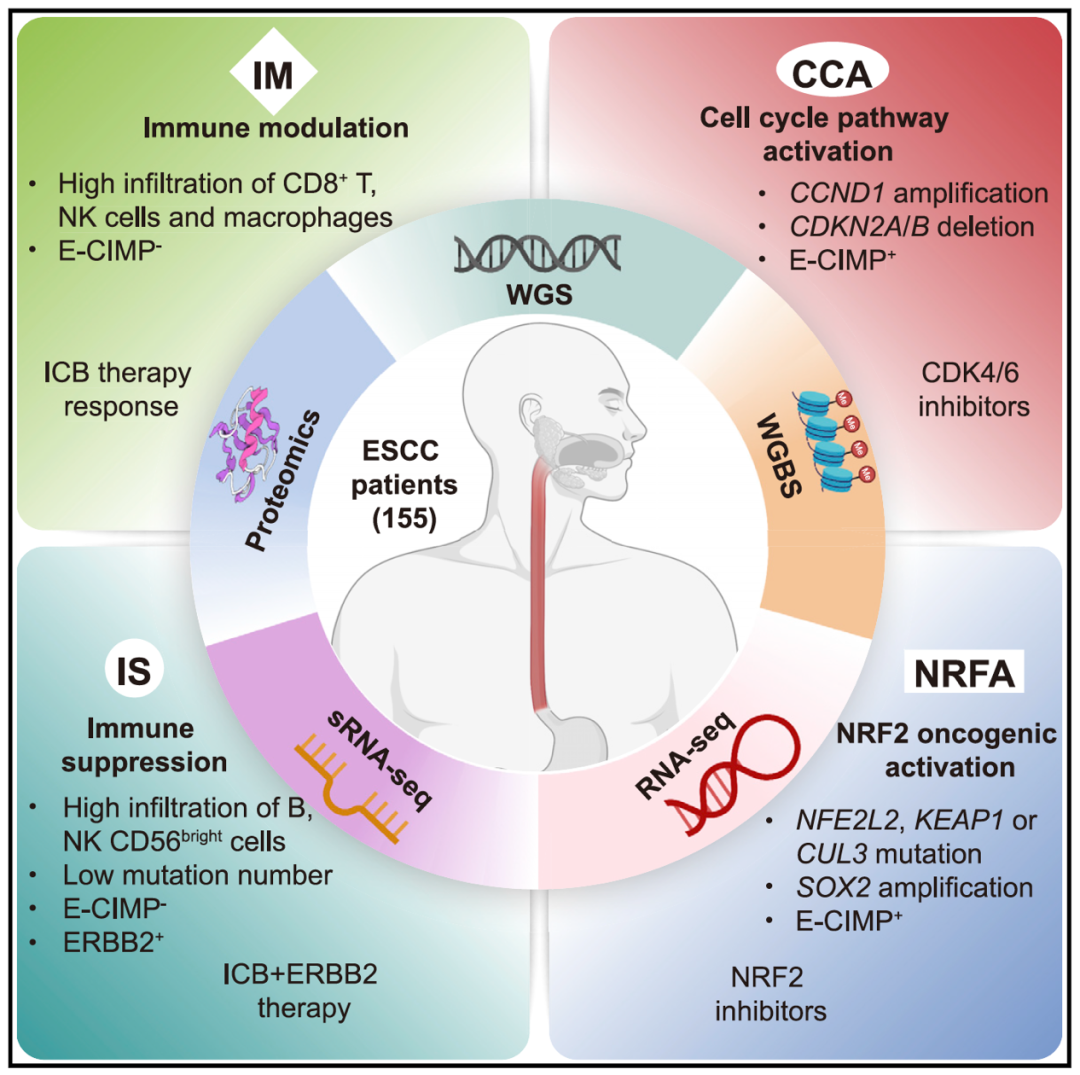
图1. 食管鳞癌多组学分子四分型及其潜在治疗靶标
CCA亚型的分子特征体现在11q13.3/CCND1扩增(74.4% vs 23.3%,CCA亚型 vs 非CCA亚型)和CDKN2A/B同源缺失(59.0% vs 37.9%,CCA亚型 vs 非CCA亚型)。其中,84.6%的CCA亚型患者有细胞周期检查点基因变异。为检测CCA亚型患者能否从CDK4/6抑制剂(palbociclib)治疗中获益,研究者构建了3类食管鳞癌类器官模型(PDOs):CDKN2A缺失(PDO4)、CDKN2A缺失/CCND1扩增(PDO7)、两个基因野生型类器官(PDO2)。研究者利用PDO半数抑制浓度(IC50)值来评估palbociclib药物敏感性,结果发现palbociclib在PDO2中的IC50(41.33μM)显著高于PDO4和PDO7(IC50分别是13.58μM和2.03μM),表明有细胞周期改变的食管鳞癌患者对palbociclib更加敏感。因此,研究者推测CCA亚型患者可从CDK4/6抑制剂的治疗中获益。
NRFA亚型的分子特征是NRF2通路基因激活,除NRF2通路基因的改变(NFE2L2扩增、NFE2L2突变、KEAP1/CUL3突变)外,研究者利用多组学数据分析发现KEAP1高甲基化和SOX2扩增,也引起NRF2通路激活。进一步通过ChIP-seq和ChIP-PCR实验证实,SOX2特异性结合NFE2L2的启动子区域,进而激活NFE2L2的转录表达。研究者对ESCC组织进行NRF2免疫组化染色(IHC)分析,结果发现NRFA亚型患者的NRF2蛋白表达水平升高,且NRFA亚型中NRF2通路基因的变异和SOX2扩增的患者NRF2蛋白表达水平最高。因此,根据多组学分析,重新定义了食管鳞癌NRFA亚型的分类标准,表明更多的患者可能从靶向NRF2的治疗中获益。
IS(免疫抑制型)和IM(免疫调节型)亚型肿瘤的特点为免疫细胞高水平浸润。但IS亚型中浸润最丰富的是B细胞和NK CD56bright细胞,其免疫微环境主要调控模块为“CD4+ Tcm-Tem”。IM亚型中浸润最丰富的为CD8+ T和巨噬细胞,其免疫微环境主要调控模块为“CD8+ T-Macrophage”。研究者通过一项抗PD-1抗体(SHR-1210)单药治疗的I期临床试验(NCT02742935),收集43名ESCC患者的样本及临床信息,患者的治疗反应被评估为部分反应(PR)、进展性疾病(PD)或稳定疾病(SD)。利用收集的肿瘤组织样本进行多组学分析,发现PR患者聚集在IM亚型,而PD患者聚集在CCA/NRFA/IS亚型。上述结果表明,ESCC的IM亚型患者可能从免疫检查点阻断治疗中获益。因此,研究者开发了由28个多组学特征分子组成的分类器,识别IM亚型患者。该分类器以85.7%的敏感性和90%的特异性,筛选抗PD-1治疗的获益者,进而预测其免疫治疗疗效,该分类器突出体现分子分型的优势和多组学检测的临床价值。
综上所述,本研究基于全基因组、表观基因组、转录组和蛋白质组的全面整合分析,首次将食管鳞癌分为CCA、NRFA、IS和IM四个分子亚型,为食管鳞癌分子分型定义了分类标准,不同亚型独特的分子特征是临床转化的“航向标”,破除了以往食管鳞癌治疗“方向模糊”的困难,有助于医学专家“对症下药”。根据基础研究数据,后续将开展针对食管鳞癌各亚型的药物研发及临床实验研究,尽早实现临床转化,制定出针对不同亚型食管鳞癌的治疗策略,让患者尽快进入食管鳞癌分子分型基础上的精准治疗时代。
原文链接:https://doi.org/10.1016/j.ccell.2022.12.004
参考文献
1. Chen W, Zheng R, Baade PD, Zhang S, Zeng H, Bray F, et al. Cancer statistics in China, 2015. CA: a cancer journal for clinicians. 2016;66(2):115-32.
2. Cui Y, Chen H, Xi R, Cui H, Zhao Y, Xu E, et al. Whole-genome sequencing of 508 patients identifies key molecular features associated with poor prognosis in esophageal squamous cell carcinoma. Cell Res.2020;30(10):902-13.
3. Lin DC, Hao JJ, Nagata Y, Xu L, Shang L, Meng X, et al. Genomic and molecular characterization of esophageal squamous cell carcinoma. Nat Genet. 2014;46(5):467-73.
4. Song Y, Li L, Ou Y, Gao Z, Li E, Li X, et al. Identification of genomic alterations in oesophageal squamous cell cancer. Nature. 2014;509(7498):91-5.
5. Li Y, Yang B, Ma Y, Peng X, Wang Z, Sheng B, et al. Phosphoproteomics reveals therapeutic targets of esophageal squamous cell carcinoma. Signal Transduct Target Ther.2021;6(1):381.
6. Cancer Genome Atlas Research N, Analysis Working Group: Asan U, Agency BCC, Brigham, Women's H, Broad I, et al. Integrated genomic characterization of oesophageal carcinoma. Nature.2017;541(7636):169-75.
7. Zhihua Liu Z, Yahui Zhao, Pengzhou Kong, Yuhao Liu, Jing Huang, Enwei Xu Wenqing Wei, Guangyu Li, Xiaolong Cheng, Liyan Xue, Yi Li, Hongyan Chen, Shuqing Wei, Ruifang Sun, Heyang Cui, Yongsheng Meng, Meilin Liu, Yang Li, Riyue Feng, Xiao Yu, Rui Zhu, Yenan Wu, Lei Li, Bin Yang, Yanchun Ma, Jiawei Wang, Wenjie Zhu, Dongjie Deng, Yanfeng Xi, Fang Wang, Hongyi Li, Shiping Guo, Xiaofei Zhuang, Xiaoyue Wang, Yuchen Jiao, Yongping Cui, Qimin Zhan. Integrated Multi-omics Profiling Yields a Clinically-Relevant Molecular Classification for Esophageal Squamous Cell Carcinoma. Cancer Cell.2023;41(1):15.